MRM Insights : Mesurer le renouvellement des protéines dans les organoïdes cérébraux dérivés d’IPSC

Jerry Dong

Jean-François Trempe
Chaque mois, dans les MRM Insights, un membre du Réseau MRM écrit sur les cellules souches et la médecine régénérative d’un point de vue différent. Ce mois-ci, Jean-François Trempe, Professeur agrégé au Département de pharmacologie et thérapeutique, et Jerry Dong, candidate à la maîtrise dans son laboratoire, nous parlent de comment mesurer le renouvellement des protéines dans les organoïdes cérébraux dérivés d’iPSC.
Mesurer le renouvellement des protéines dans les organoïdes cérébraux dérivés d’iPSC
Si un navire était ancré dans un port et que ses planches de bois étaient remplacées une par une jusqu’à ce qu’elles soient toutes renouvelées, serait-ce toujours le même navire? Comme le navire de Thésée, le corps humain et ses organes subissent un renouvellement constant au fur et à mesure que ses composantes cellulaires sont remplacées. Presque toutes nos cellules sont complètement renouvelées après une décennie, ce qui fait de vous une entité moléculaire entièrement nouvelle depuis votre conception. Pourtant, ce n’est que lorsque le processus d’élimination des protéines cellulaires en décomposition est harmonieusement balancé par la synthèse de nouvelles protéines fonctionnelles que nous conservons notre sentiment d’un soi immuable. En effet, la capacité du corps à recycler les protéines endommagées est essentielle au maintien de l’homéostasie et à une vie saine. Si ces protéines ne sont pas éliminées, elles peuvent s’accumuler, entraînant la production de protéines mal repliées qui peuvent acquérir des propriétés toxiques qui nuisent aux cellules [1]. Par exemple, la défaillance du protéasome, un complexe protéique essentiel au recyclage des composants de la cellule, est associée à des troubles neurodégénératifs tels que la maladie d’Alzheimer (MA), la maladie de Parkinson (MP) et la maladie de Huntington [2]. Différentes études ont également montré que l’autophagie, une autre voie cellulaire impliquée dans la dégradation des protéines et des organelles, est un régulateur clé des protéines sujettes à l’agrégation telles que l’alpha-synucléine, le TDP-43 et les protéines tau [3]. Les formes mutantes de ces protéines sont également des marqueurs classiques des maladies neurodégénératives. Par conséquent, le développement de médicaments qui peuvent moduler ces voies de dégradation afin de restaurer la dégradation de ces composants nocifs offre des possibilités immenses et passionnantes pour le traitement de ces maladies.
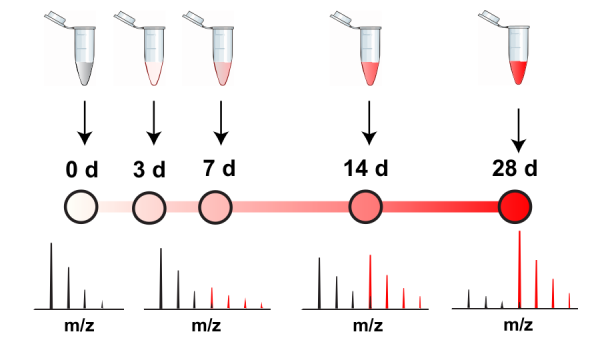
Cependant, avant de pouvoir développer des thérapies capables de réguler ces voies de dégradation, nous devons d’abord être en mesure d’identifier comment ces voies sont compromises dans un état pathologique. Cela nous oblige à mesurer le renouvellement des protéines dans les cellules saines afin qu’une ligne de base puisse être établie lors de la comparaison des taux de renouvellement avec les cellules défectueuses. Comment mesure-ton alors la dynamique des protéines dans les cellules ? Le taux de remplacement peut être mesuré à l’aide d’acides aminés marqués aux isotopes stables (SILAC) [4]. Ces acides aminés « traceurs » sont plus lourds que leurs constituants normaux dans les protéines et peuvent être détectés par spectrométrie de masse suite à leur incorporation dans des protéines nouvellement synthétisées. L’expérience commence avec des cellules ou des animaux qui sont d’abord nourris avec des acides aminés normaux (« légers »). Les cellules ou les animaux sont alors exposés à des nutriments enrichis en acides aminés « lourds ». Nous pouvons alors mesurer la vitesse à laquelle le traceur est incorporé dans des protéines spécifiques pour mesurer leurs demi-vies.
La figure 1 suivante est un schéma simplifié qui illustre ce concept. Imaginez une cellule avec des peptides appartenant aux protéines X et Y. Pendant 28 jours, les cellules seront exposées à un excès du traceur SILAC, une forme marquée de l’acide aminé leucine dans ce cas-ci. Les protéines X et Y incorporeront le traceur au fil du temps et nous pouvons mesurer le pourcentage de peptides qui ont incorporé le traceur en utilisant la spectrométrie de masse. Nous pouvons voir que la protéine Y est transformée plus rapidement que la protéine X car elle atteint un pourcentage de traceur plus rapidement. À partir des pourcentages de traceurs, nous pouvons également calculer une demi-vie pour chaque protéine donnée.
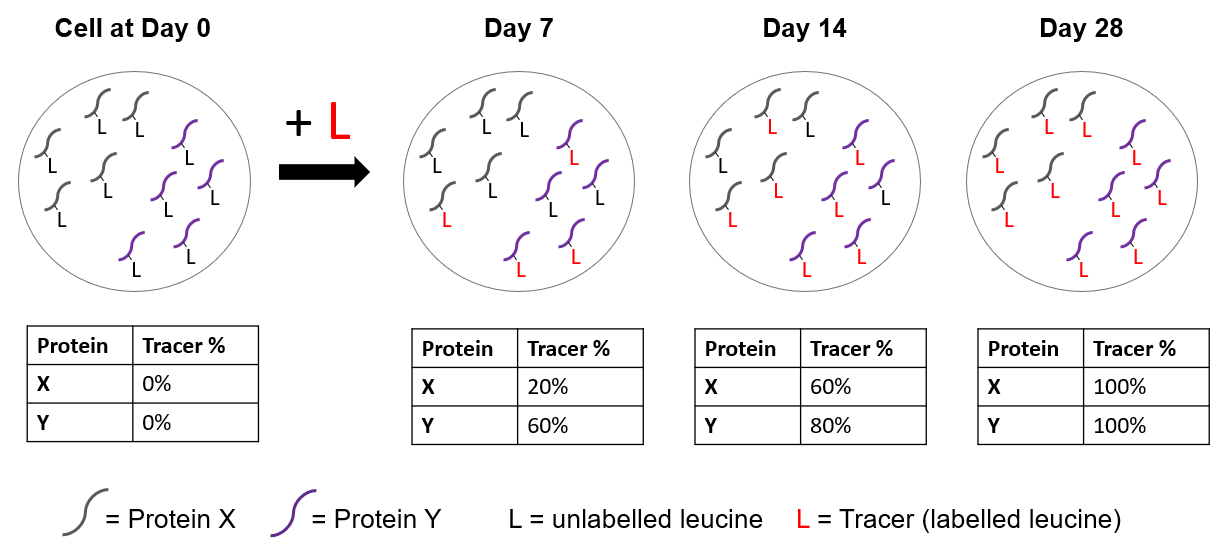
Figure 1 : Schéma démontrant deux protéines théoriques avec des demi-vies différentes incorporant le traceur marqué à des moments distincts.
Il faut être prudent dans l’interprétation de résultats tels que décrits ci-haut. Tout d’abord, nous partons du principe que les cellules ou les animaux sont dans un état stable tout au long de l’expérience de marquage (c’est-à-dire que l’organisme ne se développe pas en continu). À l’état d’équilibre, le taux de synthèse des protéines est égal au taux de dégradation des protéines et, par conséquent, le niveau global des protéines reste inchangé. Deuxièmement, lorsqu’un traceur est introduit pour la première fois dans une cellule, il est dilué par des acides aminés non marqués trouvés sous forme libre ou générés par la dégradation de protéines préexistantes [5]. Par conséquent, le bassin d’acides aminés contribuant à la formation de protéine nouvellement synthétisée contient un mélange d’acides aminés non marqués et marqués qui doivent être pris en compte dans l’estimation du taux de renouvellement. Pour plus d’informations sur la façon d’analyser ces résultats, nous, ainsi que d’autres, avons publié des protocoles pour mesurer le renouvellement des protéines à partir de telles expériences de marquage avec des données de spectrométrie de masse [5-7].
De quelle manière ces méthodes peuvent-elles se traduire en découvertes tangibles ou en traitements pour les patients ? En croisant ces techniques utilisées pour mesurer le renouvellement des protéines avec les capacités de modélisation des maladies des cellules souches, nous pouvons faire la lumière sur les mécanismes sous-jacents des troubles qui échappaient auparavant à la communauté scientifique. Par exemple, les organoïdes, des systèmes de culture tridimensionnels auto-organisés dérivés de cellules souches humaines pluripotentes et induites (iPSC), sont des outils prometteurs pour refléter des maladies [8]. Leur capacité à imiter l’architecture et la complexité d’un organe humain avec des détails remarquables s’étend au-delà des cultures cellulaires in vitro bidimensionnelles conventionnelles et des modèles animaux. Par exemple, les plaques amyloïdes-β et les enchevêtrements neurofibrillaires sont des caractéristiques pathologiques de la MA qui ne sont pas entièrement retrouvées dans les neurones humains dérivés de patients atteints de MA [9]. Les organoïdes neuronaux humains avec des mutations familiales de la MA servent à combler cette lacune, car des études ont démontré que ces systèmes de culture 3D présentent des caractéristiques clés telles que l’amyloïde-β et la pathologie tau [9]. De même, les organoïdes de type mésencéphale (MBO) reproduisent les principales caractéristiques des neurones dopaminergiques de la substantia nigra, qui sont particulièrement vulnérables dans la MP [10]. Nos collègues Vi-Nguyen Mohamed et al. ici à McGill, ont démontré que l’introduction d’une mutation familiale de la MP dans les MBO mène à l’agrégation de la protéine α-synucléine, une caractéristique typique de la MP [11].
Afin d’identifier les voies de dégradation affectées par les maladies, il est également essentiel que les demi-vies des protéines dans les modèles organoïdes soient comparables à celles observées dans le cerveau des mammifères. Ainsi, notre équipe a développé un protocole robuste pour mesurer le renouvellement des protéines dans les MBO humains en utilisant une approche basée sur SILAC [6]. Nous avons comparé les demi-vies de 773 protéines dans des MBO dérivés de type sauvage ou d’un knock-out de Parkin, une ligase d’ubiquitine E3 impliquée dans le contrôle de la qualité des mitochondries. En utilisant un protocole SILAC similaire, il a été précédemment observé que les mouches des fruits avec une délétion de Parkin présentent un renouvellement plus lent dans un sous-ensemble de protéines mitochondriales impliquées dans la respiration [12]. Étonnamment, nous n’avons trouvé aucune différence majeure dans le renouvellement des protéines mitochondriales entre les hMBO de type sauvage et Parkin-KO. Cependant, un groupe de protéines que l’on retrouve généralement dans les astrocytes avait un renouvellement plus rapide dans Parkin-KO, suggérant que ces cellules non neuronales étaient activées, conformément aux observations précédentes chez des patients humains atteints de la MP. De plus, nous avons également pu comparer nos demi-vies protéiques avec des études publiées auparavant et avons constaté que les demi-vies des hMBO ressemblent davantage à celles mesurées dans le cortex cérébral de souris qu’aux neurones corticaux primaires. Dans des travaux futurs, nous avons l’intention d’analyser les hMBO avec différentes mutations familiales de la MP pour déterminer si elles perturbent le renouvellement des protéines dans les voies partagées, ce qui fournirait une base unificatrice pour comprendre pourquoi certains neurones sont plus affectés que d’autres dans la MP.
Ainsi, la mesure du renouvellement des protéines dans les organoïdes a le potentiel de percer le mystère derrière les maladies neurodégénératives. Cependant, des défis importants restent à relever pour rendre cette méthode plus largement applicable et puissante. Alors que 773 protéines peuvent sembler beaucoup, cela représente moins de 5% de l’ensemble du protéome humain. De plus, il n’est pas possible actuellement de déterminer la cellule d’origine d’une protéine identifiée donnée. Les progrès récents dans la sensibilité et la vitesse des spectromètres de masse couplés aux technologies unicellulaires nous permettront d’explorer beaucoup plus en profondeur la dynamique protéomique des cellules individuelles du système nerveux. Cela conduira à l’identification de nouvelles voies de dégradation et de protéines pouvant être ciblées pour le développement de thérapies.
Références
1. Fecto, F., Esengul, Y.T. & Siddique, T. (2014). Protein recycling pathways in neurodegenerative diseases. Alz Res Therapy 6, 13. https://doi.org/10.1186/alzrt243
2. Zheng, Q., Huang, T., Zhang, L., Zhou, Y., Luo, H., Xu, H., & Wang, X. (2016). Dysregulation of Ubiquitin-Proteasome System in Neurodegenerative Diseases. Frontiers in aging neuroscience, 8, 303. https://doi.org/10.3389/fnagi.2016.00303
3. Frake, R. A., Ricketts, T., Menzies, F. M., & Rubinsztein, D. C. (2015). Autophagy and neurodegeneration. The Journal of clinical investigation, 125(1), 65–74. https://doi.org/10.1172/JCI73944
4. Ross, A. B., Langer, J. D., & Jovanovic, M. (2021). Proteome Turnover in the Spotlight: Approaches, Applications, and Perspectives. Molecular & cellular proteomics : MCP, 20, 100016. https://doi.org/10.1074/mcp.R120.002190
5. Hsieh, E. J., Shulman, N. J., Dai, D. F., Vincow, E. S., Karunadharma, P. P., Pallanck, L., Rabinovitch, P. S., & MacCoss, M. J. (2012). Topograph, a software platform for precursor enrichment corrected global protein turnover measurements. Molecular & cellular proteomics : MCP, 11(11), 1468–1474. https://doi.org/10.1074/mcp.O112.017699
6. Dong, J., Duchesne, A., Bayne, A. N., Mohamed, N. V., Yi, W., Mathur, M., Chen, C., You, Z., Abdian, N., Taylor, L., Fon, E. A., Durcan, T. M., & Trempe, J. F. (2022). An approach to measuring protein turnover in human induced pluripotent stem cell organoids by mass spectrometry. Methods (San Diego, Calif.), 203, 17–27. https://doi.org/10.1016/j.ymeth.2022.03.011
7. Price, J. C., Guan, S., Burlingame, A., Prusiner, S. B., & Ghaemmaghami, S. (2010). Analysis of proteome dynamics in the mouse brain. Proceedings of the National Academy of Sciences of the United States of America, 107(32), 14508–14513. https://doi.org/10.1073/pnas.1006551107
8. Kim, J., Koo, B. K., & Knoblich, J. A. (2020). Human organoids: model systems for human biology and medicine. Nature reviews. Molecular cell biology, 21(10), 571–584. https://doi.org/10.1038/s41580-020-0259-3
9. Choi, S. H., Kim, Y. H., Hebisch, M., Sliwinski, C., Lee, S., D’Avanzo, C., Chen, H., Hooli, B., Asselin, C., Muffat, J., Klee, J. B., Zhang, C., Wainger, B. J., Peitz, M., Kovacs, D. M., Woolf, C. J., Wagner, S. L., Tanzi, R. E., & Kim, D. Y. (2014). A three-dimensional human neural cell culture model of Alzheimer’s disease. Nature, 515(7526), 274–278. https://doi.org/10.1038/nature13800
10. Jo, J.et al. (2016). Midbrain-like Organoids from Human Pluripotent Stem Cells Contain Functional Dopaminergic and Neuromelanin-Producing Neurons. Cell stem cell, 19(2), 248–257. https://doi.org/10.1016/j.stem.2016.07.005
11. Mohamed, N. V. et al. (2021). Midbrain organoids with an SNCA gene triplication model key features of synucleinopathy. Brain communications, 3(4), fcab223. https://doi.org/10.1093/braincomms/fcab223
12. Vincow, E. S., Merrihew, G., Thomas, R. E., Shulman, N. J., Beyer, R. P., MacCoss, M. J., & Pallanck, L. J. (2013). The PINK1-Parkin pathway promotes both mitophagy and selective respiratory chain turnover in vivo. Proceedings of the National Academy of Sciences of the United States of America, 110(16), 6400–6405. https://doi.org/10.1073/pnas.1221132110
